为什么要进行多重比较校正
当在同一个数据集上进行多次统计检验时,就需要进行多重比较校正。举个简单的例子,A、B两组被试,我们从每个被试身上得出10个指标。如果我们要研究A、B两组被试的某一个指标是否存在显著差异,那么此时我们只做一次统计分析就行;假设这个指标的p值小于0.05,我们会认为这个指标在A、B两组之间存在显著差异,此时,我们犯错的概率(或者称为假阳性率)是5%。假设我们把这10个指标都进行了统计分析,即使每个独立的指标的p值都小于0.05,此时我们犯错的概率不再是5%,至少有一个false positive的概率是1-(0.95)^10=0.4013,也就是说此时我们犯错的概率达到40%多,这在统计学上是不可接受的。因此,需要进行多重比较校正。
因为假设检验是对原假设有利。(对,因为它本质上是一个反证法)。思想:
- 先预设原假设是成立的
- 那么看看当前的情况在原假设的条件下是不是极小概率事件,比如发生概率<5%
- 如果是的话。那原假设就很可疑,迫不得已认为它不成立
- 不是的话。那原假设就凑活着用吧
- 如果你多次使用假设检验,还好死不死全都通过了的话!
你的逻辑链条里将出现大量“就凑活着用吧”的命题,当然不靠谱啦!
矫正方法
在多重假设检验中,有不同的控制指标,包括:
- Per comparison error rate (PCER, 比较误差率): Type I Error的期望数目比总假设数
- Family-wise error rate (FWER): 至少出现1次Type I Error
- False discovery rate (FDR, 错误发现率): Type I Error在总拒绝数量中的比率(错误拒绝率)
- Positive false discovery rate (pFDR, 阳性错误发现率): 错误发现的比率
在实际应用中,我们一般希望减少Type I Error出现(错误拒绝H0)的可能,因此主要考虑FWER和FDR这两个数据。FWER指标有Bonferroni 校正和Holm’s方法;FDR指标有Benjamin and Hochberg (BH)方法。
基于Bonferroni 法的校正过程
算法
Family-wise error rate :
c为总检验次数(即P值的个数); αPC为每次比较中的错误率(通常为0.05)。所以在3次比较中,αFW的值为 1-(1-0.05)^3 = 0.143。
有两种方法进行矫正(如下图):
- Approach 1: 将原始的p-value和 αPC /3 = 0.05/3 = 0.017 比较,原始P value小于 矫正后的p-value(如0.017)即可认为是显著差异的p-value;
- Approach 2:将原始的p-value和总检验次数(如3)相乘,得到矫正后的p-value,如果矫正后的p-value可认为是显著差异的p-value;

如果原始的P值为0.05,检验次数为10000次,那么在Bonferroni 校正中,校正的阈值就等于5%/ 10000 = 0.000005,所有P值超过0.00005的结果都被认为是不可靠的。这样的话假阳性结果在10000次检验中出现的次数为 10000 * 0.000005 =0.5,还不到1次。
优缺点
Bonferroni 校正法可以称作是“最简单粗暴有效”的校正方法,它拒绝了所有的假阳性结果发生的可能性,通过对p值的阈值进行校正来实现消除假阳性结果。但是这也存在问题:Bonferroni 委实太过严格,被校正后的阈值拒绝的不只有假阳性结果,很多阳性结果也会被它拒绝。
R实现
1 | p=c(0.053,0.001,0.045,0.03,0.02,0.01) |
基于BH法的FDR校正过程
算法
举个例子,我们最开始设定的情况中进行了10000次检验,这次我们设定FDR在一定的范围,如FDR<0.05,如果我们的检验对象为差异表达的基因,那么在10000次检验中假如得到了500个基因,那么这500个基因中的假阳性结果小于 500*5% = 25 个。比较常用的BH发算法如下:
- BH 法需要将总计m次检验的结果按由小到大进行排序,k为其中一次检验结果的P值所对应的排名。
- 找到符合原始阈值α的最大的k值,满足P(k)<=α*k/m,认为排名从1到k的所有检验存在显著差异,并计算对应的q值公式为q = p*(m/k)。
举个例子,如果我们有总共六个结果进行FDR校正:
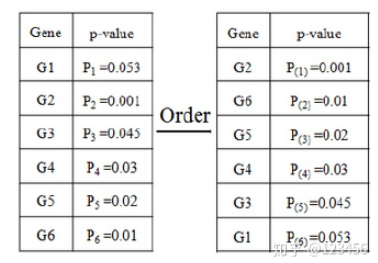
按α=0.05进行计算:
排名第四的 P (4) = 0.03 < 0.05*4/6 = 0.033,符合要求
排名第五的 P (5)= 0.045 > 0.05*5/6 = 0.041,不满足P(k)<=α*k/m,因此在这个列表里排名前四的G2,G6,G5,G4 为具有显著差异的基因。
我们也可以用q值进行FDR校正:
q-value = P * (k/m)
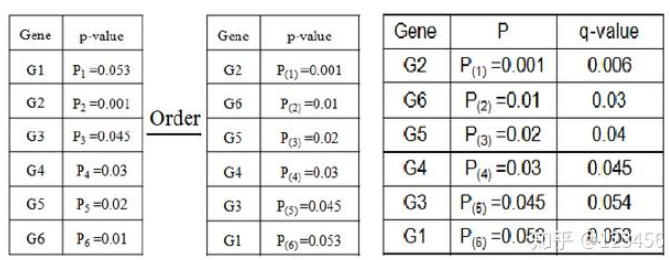
排名第五的G3,其q值大于0.05,故G2,G6,G5,G4 为具有显著差异的基因。
优缺点
相对Bonferroni 来说,FDR温和得多,这种校正方法不追求完全没有假阳性结果,而是将假阳性结果和真阳性的比例控制在一定范围内。
R实现
1 | p=c(0.053,0.001,0.045,0.03,0.02,0.01) |
引用
谈一谈两种常用的多重比较校正方法
多重检验,P值校正
多重假设检验及其生物学应用
多重假设检验:Bonferroni 和 FDR
how2stats - What is the Bonferroni Correction?
StatQuest - False Discovery Rates, FDR, clearly explained
参考文章如引起任何侵权问题,可以与我联系,谢谢。